2022.3.8
接手蔡姐的鹅膏属数据,其中他从网上下载的Amanita muscaria的数据被我换成了从NCBI上下载的已经注释好的,剩下的10号标本她给的。
分析codes
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
| ls>list.txt
list=$(cat list.txt)
for i in $list;do
cd ${i};
fastp -i ${i}_1.clean.fq.gz -o ${i}_R1.fq.gz -I ${i}_2.clean.fq.gz -O ${i}_R2.fq.gz -w 16;
cd ..;
done
for i in $list;do
cd ${i};
time spades -o spades_result -1 ${i}_R1.fq.gz -2 ${i}_R2.fq.gz -t 20;
cd ..;
done
conda activate buscoEnv
for i in $list;do
cd ${i};
time busco -i ./spades_result/contigs.fasta -o busco -m geno -c 20 -l ~/database/BUSCO/agaricales_odb10;
cd ..;
done
dos2unix list2.txt
list=$(cat list.txt)
list2=$(cat list2.txt)
for i in $list2;do
echo -ne "${i}\t" >> test.txt;
find ./*/busco/run_agaricales_odb10/busco_sequences/single_copy_busco_sequences -name "${i}.faa"| wc -l >> genespe.txt;
done
dos2unix list3.txt
list3=$(cat list3.txt)
for i in $list3;do
for j in $list;do
echo ">${j}" >> ../all/${i}.fasta;
grep -v ">" ../${j}/busco/run_agaricales_odb10/busco_sequences/single_copy_busco_sequences/${i}.faa >> ../all/${i}.fasta;
done
done
for i in *.fasta;do
linsi --thread 20 ${i} > ${i}.mafft;
done
for i in *.mafft;do
Gblocks $i -b4=2 -b5=h -t=p;
done
for i in *.mafft-gb;do
seqkit sort $i > $i.sort;
done
for i in *.sort;do
seqkit seq $i -w 0 > $i.seqkit;
done
paste -d " " *.seqkit > all.fa
cd
java -jar prottest-3.4-2/prottest-3.4.2.jar -i /mnt/d/Amanita/all/all.fa -all-distributions -F -AIC -BIC -tc 0.5 -threads 20 -o /mnt/d/Amanita/all/prottest.out
raxmlHPC-PTHREADS-SSE3 -T 20 -f a -x 123 -p 123 -N 1000 -m PROTGAMMAIJTTF -k -O -n all.tre -s all.fa
iqtree -s all.fa -m TEST -nt 20
iqtree -s all.fa -m LG+F+I+G4 -bb 1000 -nt 20
iqtree -s all.fa -m MFP -bb 1000 -nt 20
|
组装结果
| NumSpe |
contigs(Mb) |
| Q1 |
77.5 |
| Q4 |
123 |
| Q5 |
51 |
| Q6 |
64.9 |
| Q7 |
51.6 |
| Q8 |
91.1 |
| Q9 |
70.4 |
| Q10 |
46.3 |
| Q11 |
119 |
 |
|
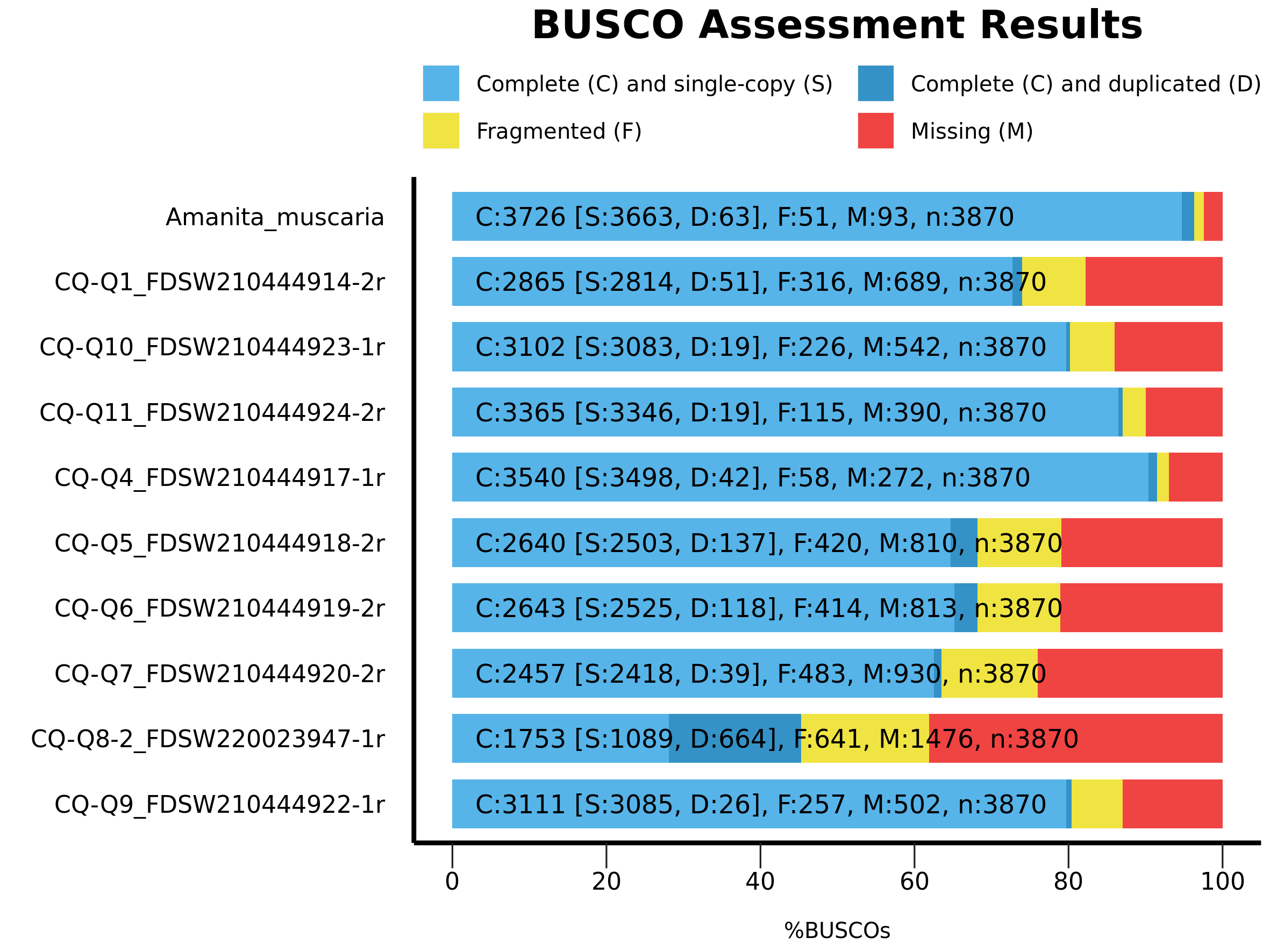
| 除了Q8的单拷贝提取结果比较差以外,其他的都还不错。 |
|
建树结果
这里就不展示了!

